Genetic manipulation of gut microbes enables single-gene interrogation in a complex microbiome
为什么读这篇文章
- 今天读完Chunjun Guo的2019年的Science文章,趁热打铁看一下今年的cell文章
- 思考有没有可能将代谢组学应用到相关方面的可能
- 学习和了解为主
- BioArt
Introduction
- 多组学方法已经发行了很多菌群基因与宿主biology相关,但其因果机制难以阐释,主要因为很多来自于非模式的菌株(如:厚壁菌门Firmicutes,梭菌Clostridia),此外这些菌株是没有被基因组测序(理论上这个不应该是简单的吗),该如何引入外源基因以及选择哪个工具都是未知的。
- 目前没有一个方法,能够在没有基因组先验知识的背景下,能够进行基因转移并构建基因操作体系
- 这样的流程将有3个好处:
- 很多多组学显示出显著性关联的gene都只特定地表达在非模式菌株中。可以帮助揭示上述的因果关系
- 已知gut microbiota发挥了重要调节作用,但是哪个菌和基因起作用却知之甚少。这个方法可以将先前无法靶标的基因变成可能
- 揭示分子机制以及基因编辑用于菌群治疗
- 本文的主要特点:
- 鉴定了88个菌株(绝大多数都是非模式菌株)的外源基因转入方法以及遗传操作的工具。(请教了王博士,其实质粒sgRNA的转入就是外源基因的)
An overview of the GM pipeline
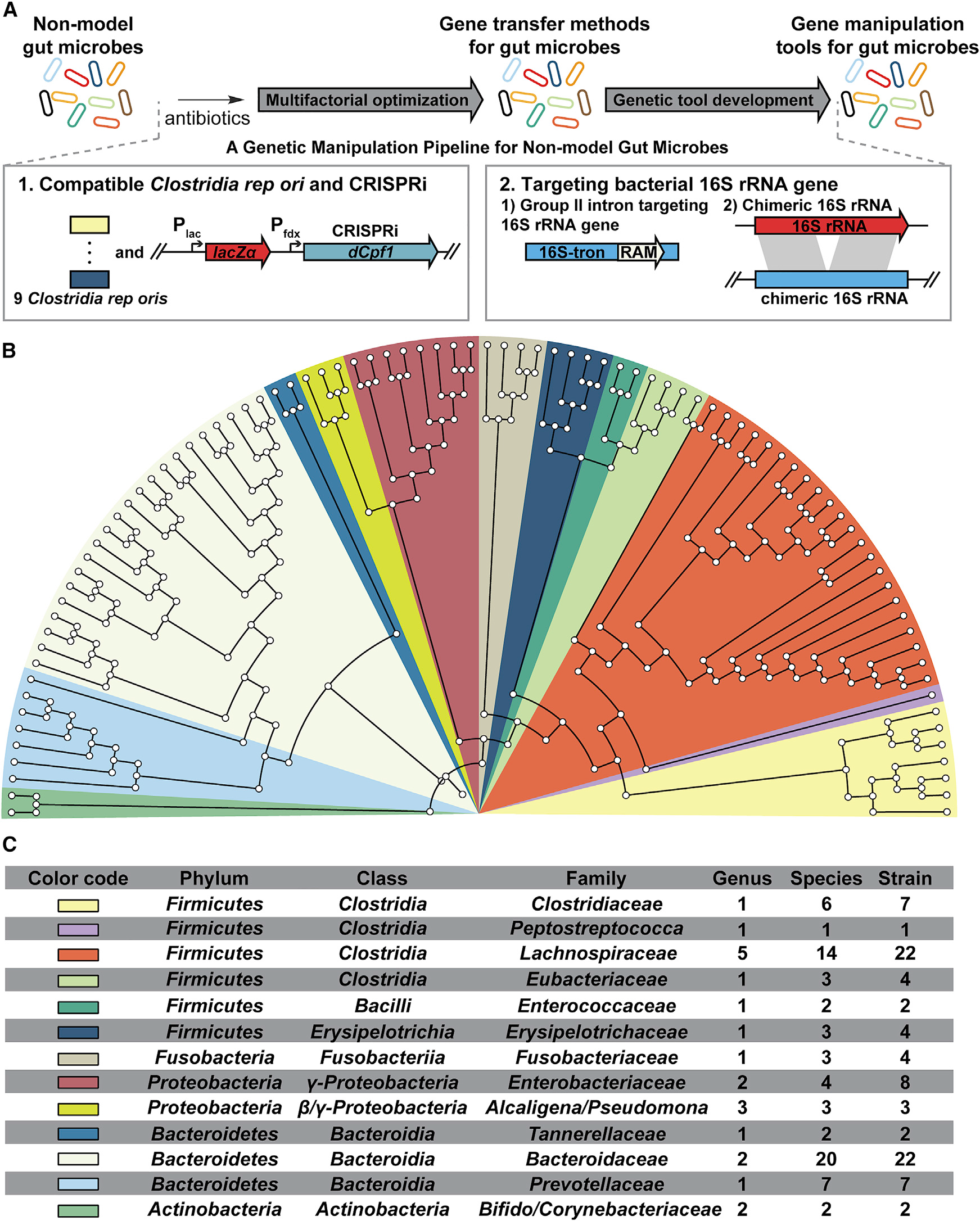
总览,面临的主要困难:
- 没有之前报道过的抗生素makers可以通用地应用于nonmodel microbes
- 对于厚壁菌和梭菌这些高丰度的非模式菌,没有遗传工具可以使用
- 许多isolated没有被完整测序
Selection of gut microbes and screening their culture conditions and antibiotic resistance
选择合适的菌株和抗生素:
- 抗生素的最低抑制浓度是允许的(minimal inhibitory concerntration, MIC)
- 能够有抑制作用但是对E. coli有活性
A multifactorial optimization to identify gene transfer methodology for nonmodel Clostridia
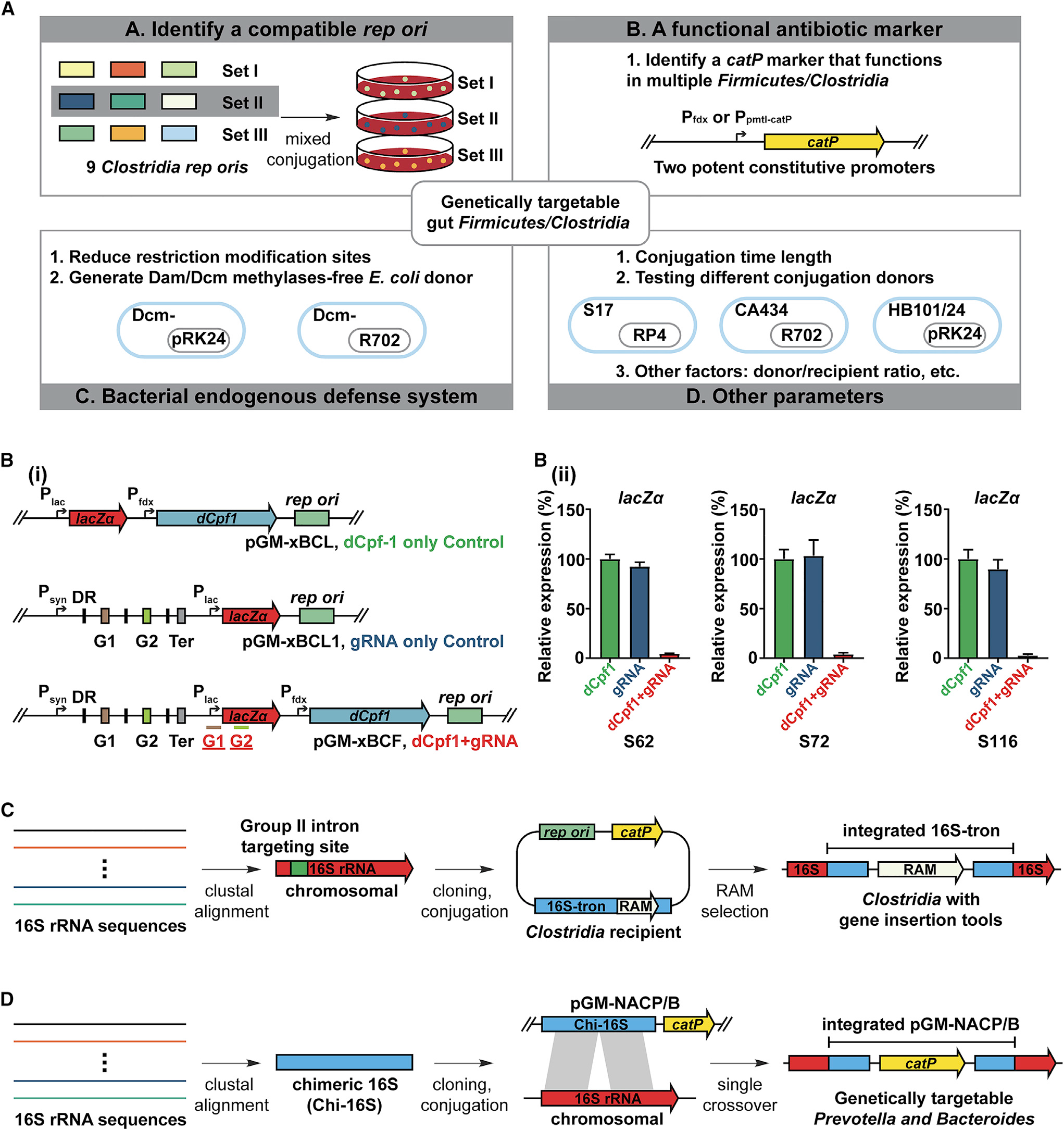
- 进行了一系列的优化,具体细节不太能看懂,包括了
- catP maker,Ppmty-catp以及Pfdx-cs可以引入抗生素抗性
- 使用了E.coli Dcm methylase-free “sExpress” conjugation donor
- 一些参数的优化
- 结果:拥有41%的成功率,表明发展这种发展的可能性
Testing CRISPRi-dCpf1 system in multiple Clostridia commensals
- 选择了CRISPRi-dCpf1: 不会导致双链的切断,低毒性以及高的接和效率。
- 通过敲除clostridia普遍存在中的lacZa,证明了这个方法能够普遍适用于梭菌 (Fig 2b)
A strategy targeting bacterial 16S rRNA genes to generate targeted gene insertion tools in nonmodel gut commensals
Constructing mutants to modulate Clostridia gene transcription and microbiome metabolites production
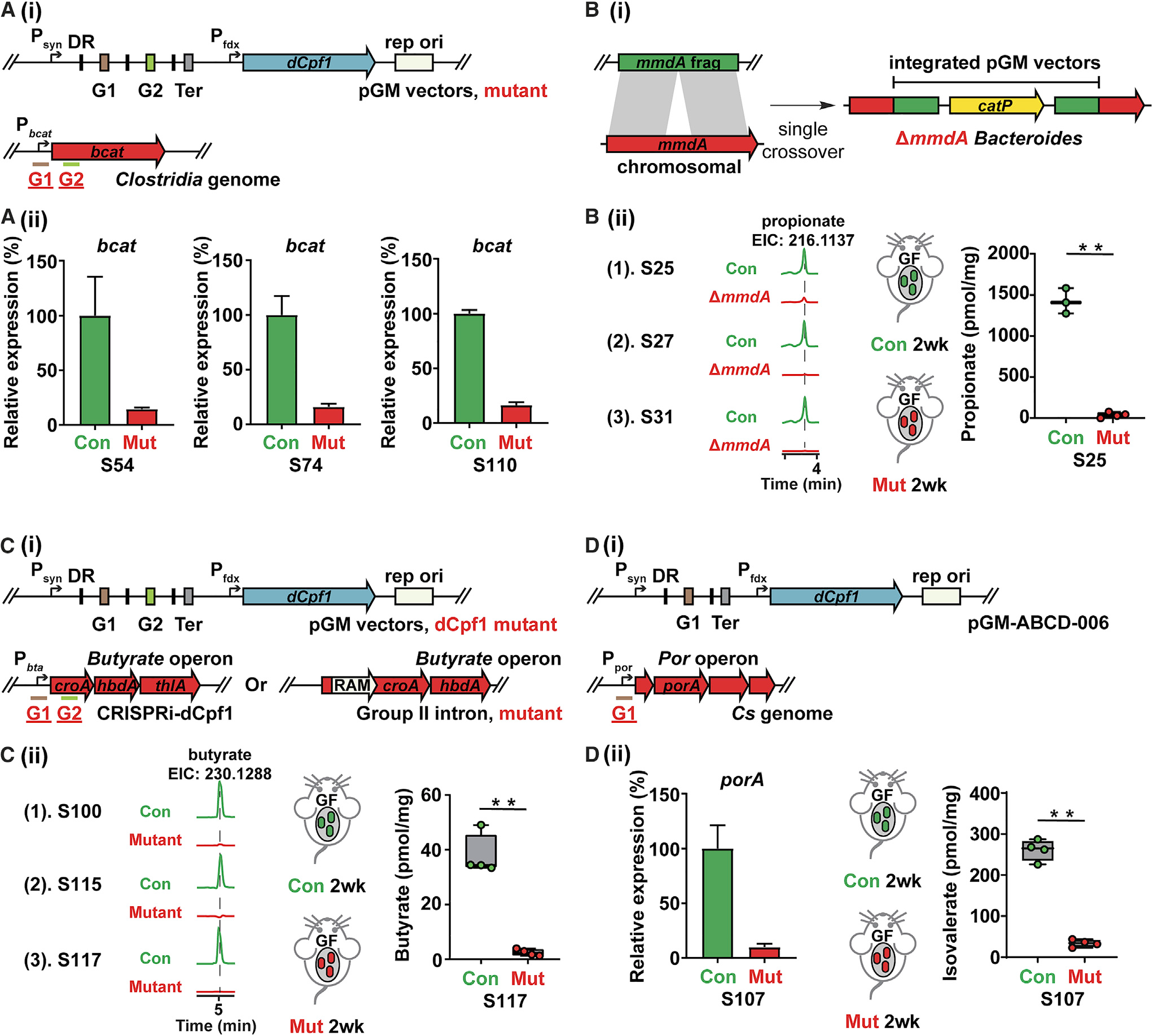
- 这个部分就和science那篇文章比较像了
- 选择abundant producers,敲了SCFAs (mmdA, croA),BSCFAs(porA)的一系列已知基因,证明这个方法可以用来调控代MDM
A case study of Clostridia-specific bile acid 7a-dehydroxylation
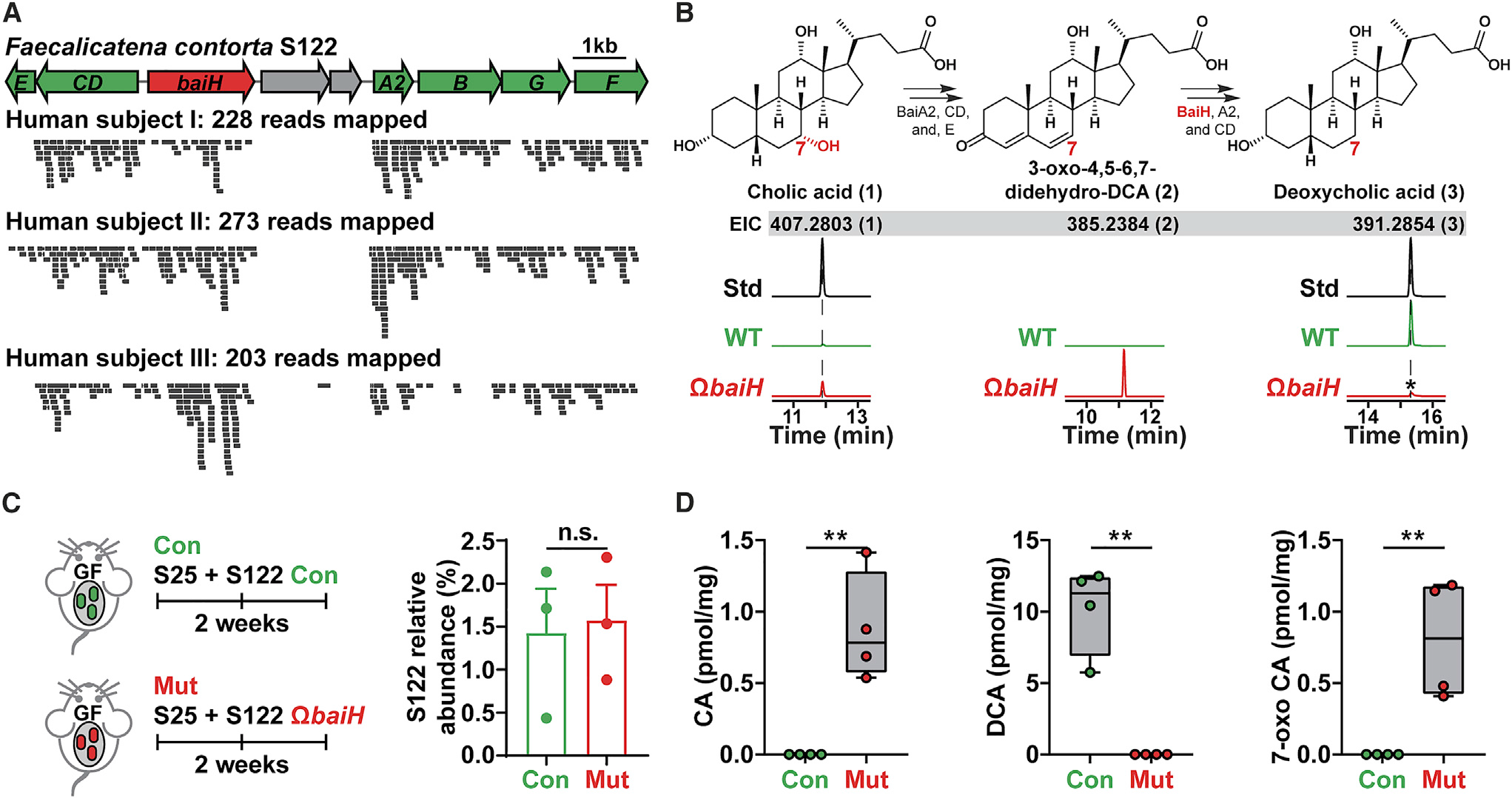
- 选择这个通路的原因:
- 发现是122菌株可以高效地转换CA/CDCA,以及DCA/LCA
- DCA/LCA及其衍生化是宿主host secondary bile acid的主要组成
- 两性的bile acid具特殊的生物学功能
-
通过生信分析以及发现潜在的bai外显子表明:
- 对S122进行测序并发现bai外显子在两个人群队列中大规模存在
- 和其他的7a脱羟基的比起来虽然基因组上丰度同样比较低,但宿主的肠道中却高度转录
- 比C. hiranonis 和C. scindens更普遍
-
结果:
- 体外突变成功,DCA明显下降,中间产物累积
- 但是定植小鼠不成功,但发现可以和S25菌株一起定植(这个怎么发现的没讲),一起定植后就成功了
The baiH gene has significant effects on the host bile acid pool and microbiota composition
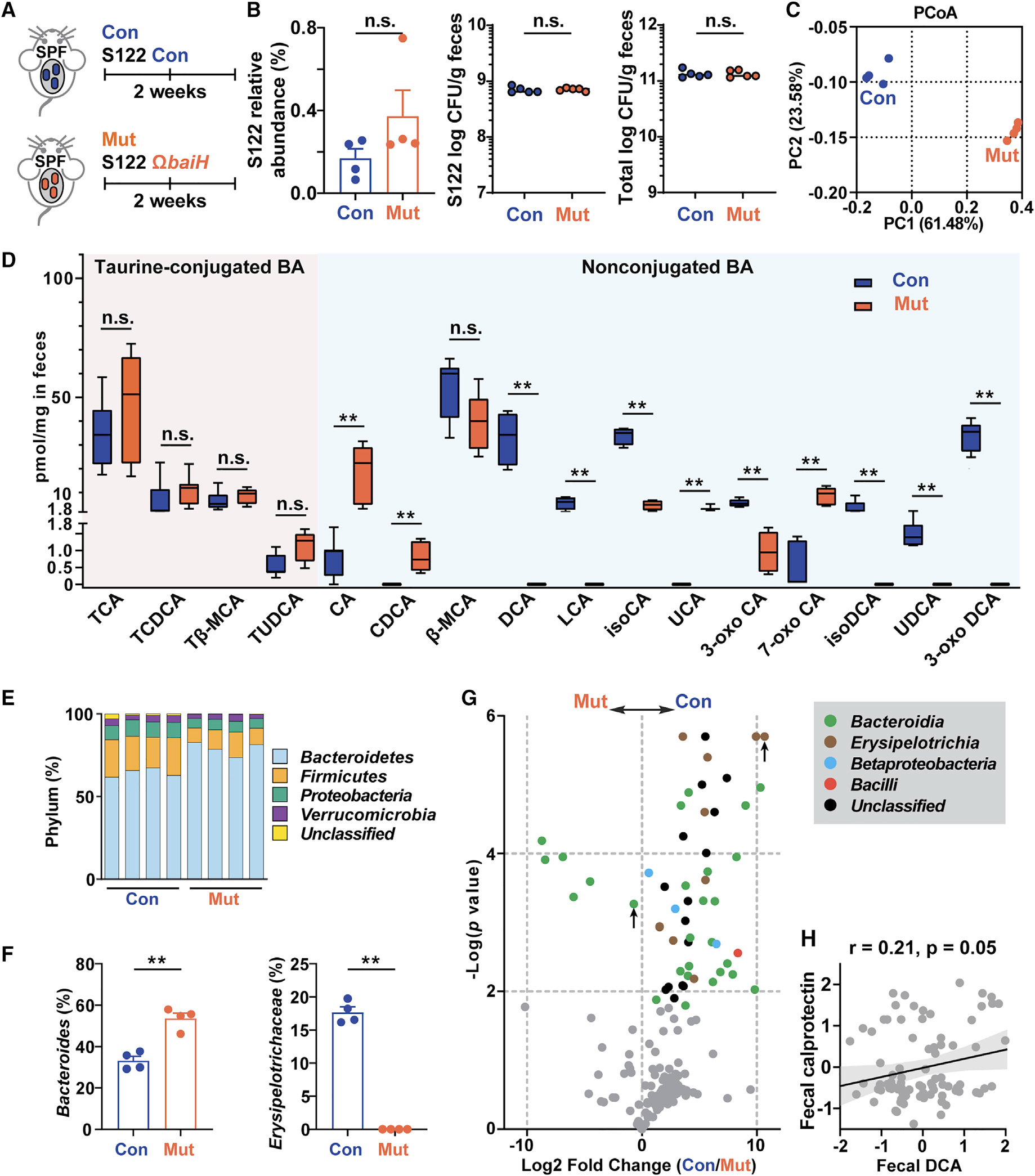
- 在复杂的菌群体系(如SPF小鼠)中敲除baiH基因,但这个不好实现。正如作者所说,这SPF小鼠中已经有了一个复杂的菌落来进行7a脱羟活性。
- 处理方法是control和baiH突变株中同时转入thiamphenicol marker,然后在小鼠的饮用水里加入thiamphenicol (15 mg/ml) and erythromycin (10 mg/ml),从而实现相同的细菌载量 (粪便的16S rRNA sequencing)
- 下面的数据分析比较简单:
- PCoA分析表明聚类与突变显著相关
- 突变和controly有明显不同的bile acid pool;conjugated BCA没有差别,表示其不会影响修饰的活性;DCA and its derivatives在control累积
- knocking out baiH可以改变宿主microbiome的组成(这个很有意思)。
Assessing the effect of baiH on intestinal inflammation
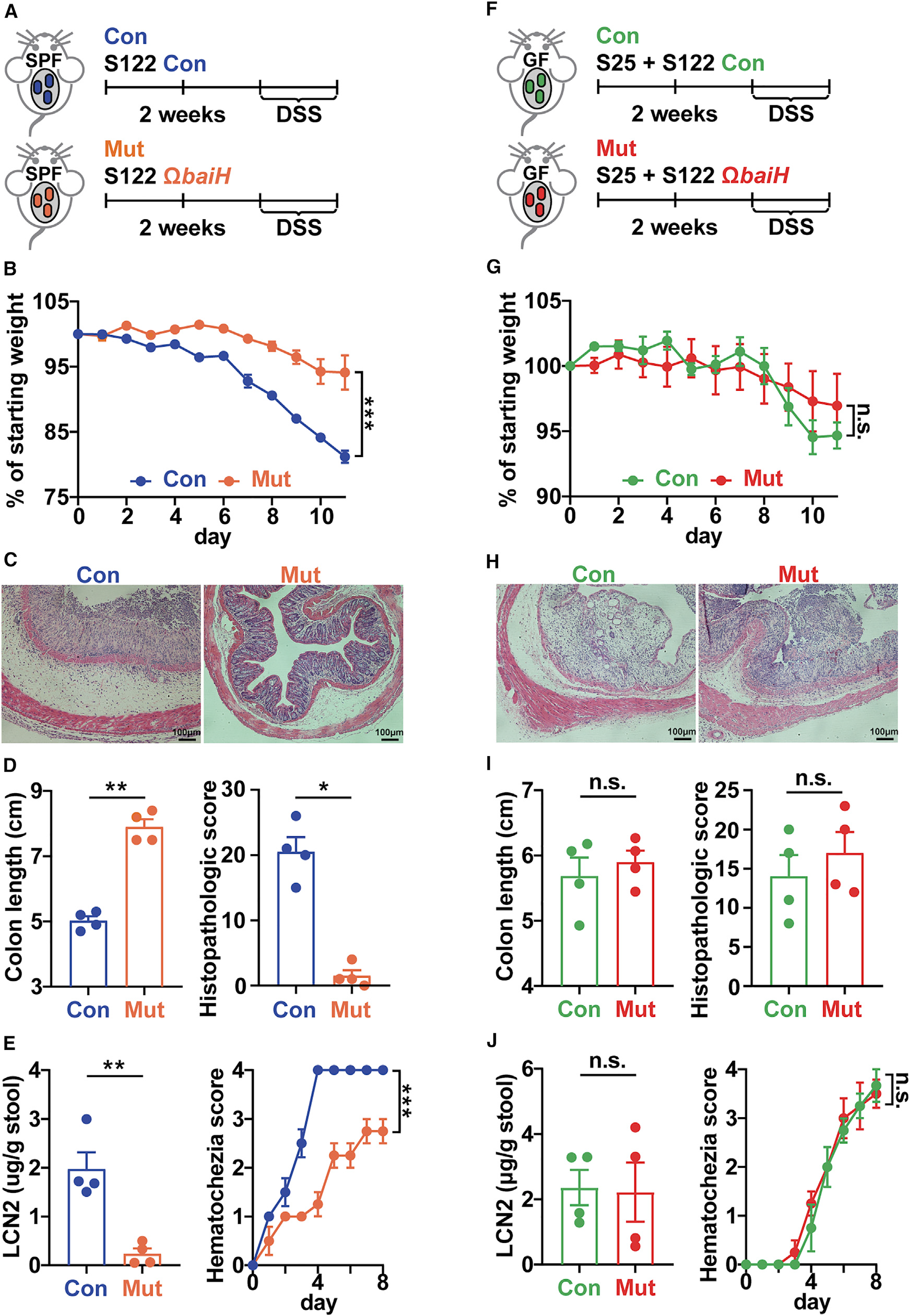
- 评估baiH在DSS诱导模型(疾病模型)中是否能够调节免疫
- 设计:
- 分别S122 control,S122 control + bai突变,肠道免疫变化明显
- 但是当加入S25之后就没有明显的变化 (这个结果也很有意思)
- 结论:baiH介导的炎症反应是依赖于微生物群的,而在复杂的微生物群中,baiH的减少会重塑宿主胆汁酸的形态,并预设肠道微生物组的组成,使其处于更有保护作用的对抗状态DSS-induced结肠炎
The baiH-mediated microbiota composition shift exacerbates DSS-induced colitis in gnotobiotic mice
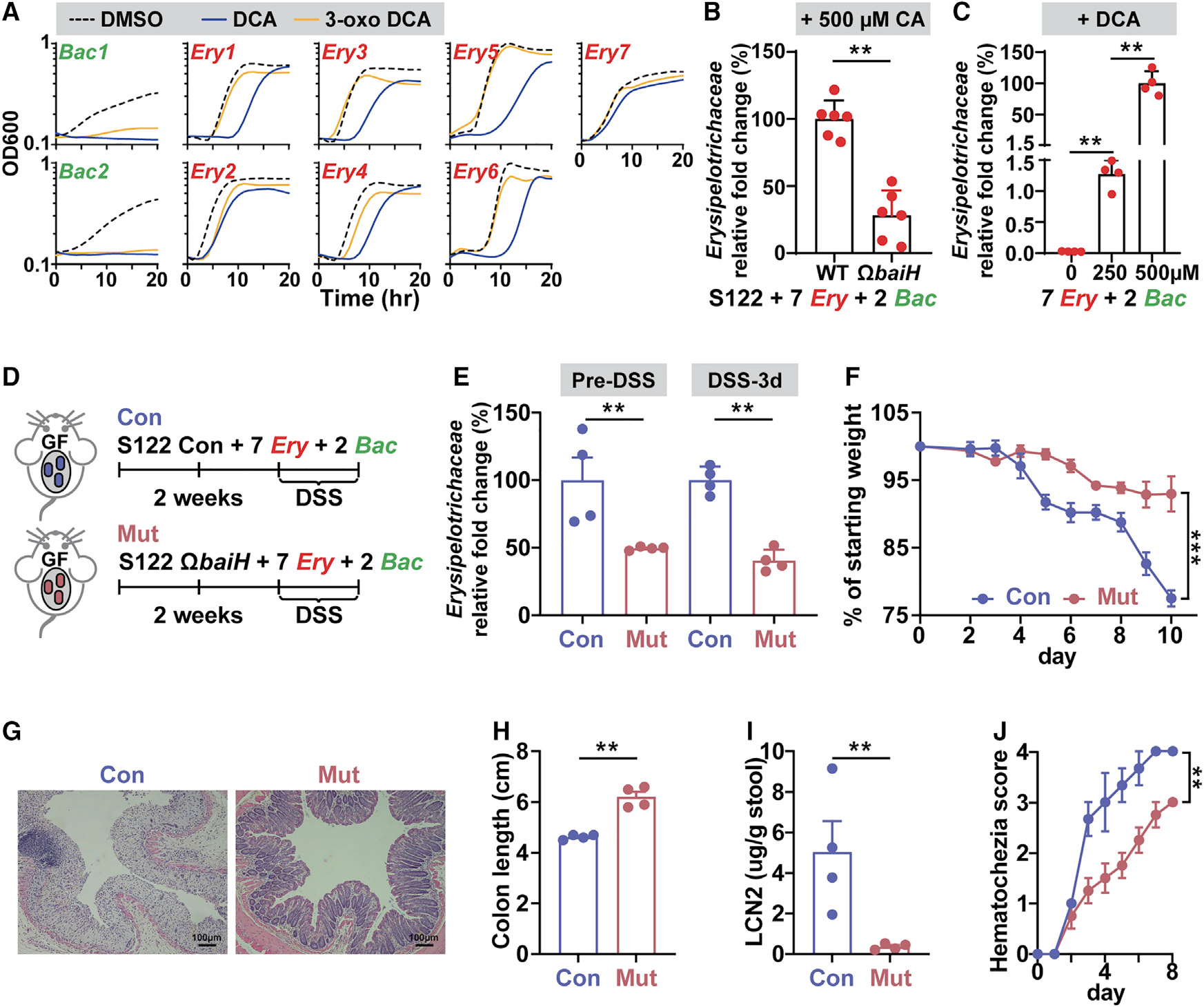
- 验证baiH突变在GF小鼠中造成的改变是否与在与DSS刺激对SPF小鼠造成的改变相关。
- baiH 是否会在体内驱动丹毒科的扩张,以及这种微生物群组成的变
感受
- 文章还是很一如既往的很有逻辑,对于做方法的人还是很好了解,即使像我这样完全没有遗传学操作的背景
- 但有一个疑问,这篇文章为了验证和展示结果,都是选择比较熟知的gene进行操作,但是真正使用的时候,这些关键gene从哪里来?(人群队列的多组学数据可能是一个来源点)
- 怎么有可能把这个方法和我熟悉的非靶向代谢组学技术联系起来?BGC那条路可能是一条;多组学数据出来的关键基因,逐个敲除,类似于文库,然后做非靶向代谢组学筛选,但这个可以回答的生物学问题是什么?