2017 Cell Discovery of Reactive Microbiota-Derived Metabolites that Inhibit Host Proteases
为什么读这篇文章
- Michael组近几年文章中另外一篇从基因组出发发现MDM代谢物的文章
- Chunjun Guo在Michael组主要工作之一,另一篇是用CRISPER-Cas9进行遗传学操作
- 这篇文章可以看做是2014年那篇Cell的延续
Background
- 菌群通过多种方式影响宿主,但是绝大多数在分子机制层面上是不清楚的
- 2014 Doina的Cell paper表明人类基因组中存在数千个功能未知的生物合成位点(BGC),还包括一些在50%以上HMP队列样本中存在的大家族
- 这些基因能够合成什么存在很大的gap,并且为发现新的meditor的调控机制提供了可能的契机
- 从之前的数据出发,去表征了nonribosomal peptide synthetase (NRPS) gene clusters,基于以下2个原因:
- 在90%以上的HMP stool样本中存在,证明其广泛存在
- 绝大部分属于gut bacterial genome sequences,只有少量environmental isolates, 表明这些BGC生成的小分子可能在肠道中起到重要的interspecies signaling作用
Computational Analysis of the Gut NRPS Cluster Family
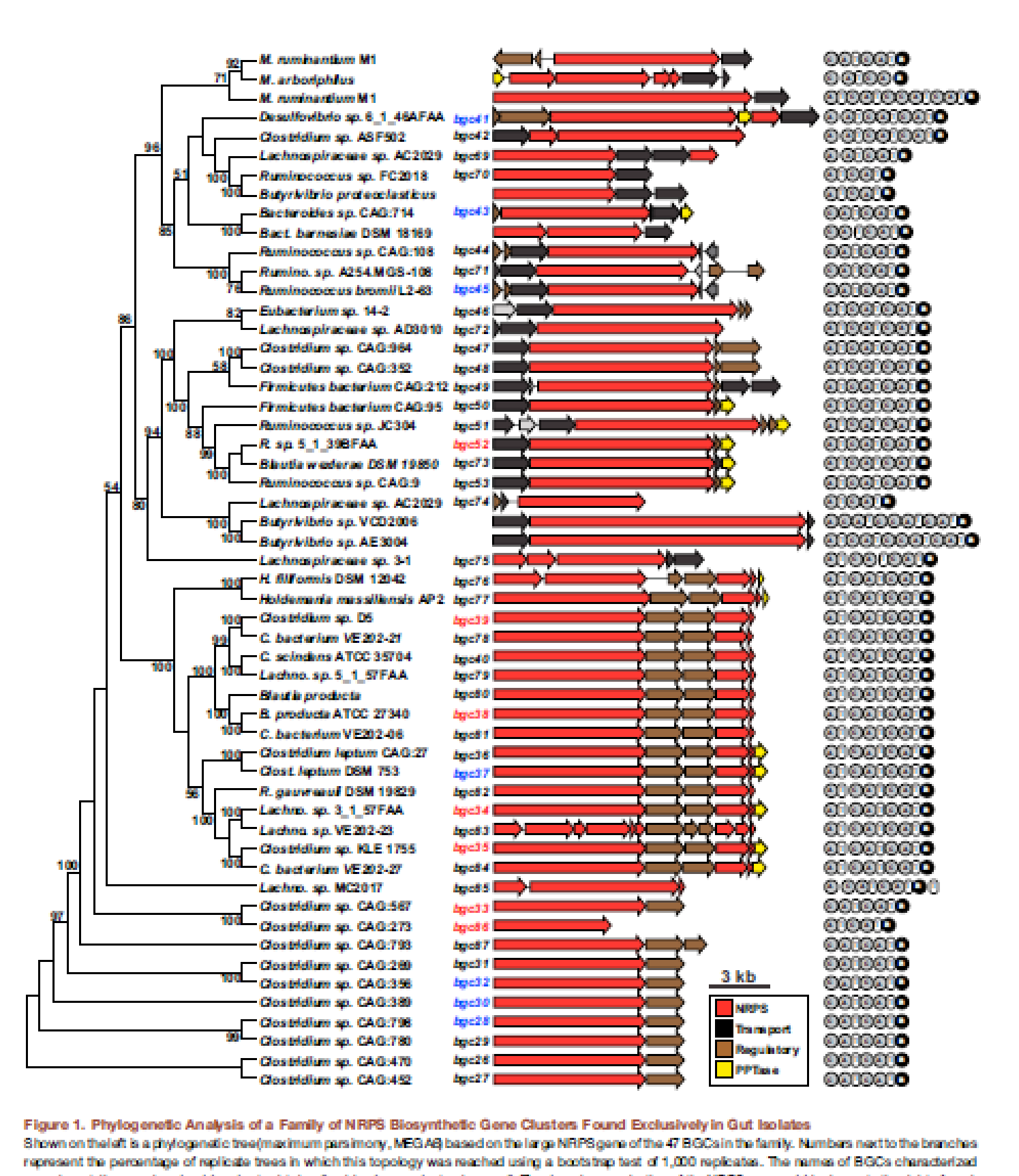
- 新发现了19个,一共有47个BGC,具有以下特点:
- 4个分支组成具备不同的特点,根据module,每一个都有终端还原酶domain
- 均来自anaerobic firmicutes(厌氧厚壁菌门)和clostridia (梭状龅牙杆菌),少数来自革兰阴性式杆菌
- 几乎全来自于人类肠道或者其他mammals
- 每个分支均包含一些没有被分离过的cluster
- 从4个分支中选了14个cluster,由于这些菌株没有被遗传学操作过,且这类clostridia对于遗传学手段有抗性,因此没有使用敲除的方式,而是进行体外表达(Ecoli和Bacillus)
Experimental Analysis of the Gut NRPS Gene Clusters

- 对上面表达的E.Coli进行LC-MS分析,然后分离纯化出其中bgc35和bgc52中的几个新峰,这些新峰是pyrazionones和dihydropyazionones
- 问题:是怎么把目标定在pyrazionones和dihydropyazionones
- 列举的几个理由:
- 220和300nm的UV吸收符合pyrazinone core
- HRMS的Mass和formula推断,α-amino-acid-derived pyrazinone和dihydropyazionones
- 1D加2D核磁确认
- 结论:7 out of 14个BGC中,鉴定到了32个化合物,其中28个是没有被报道过的
The Same Molecules Are Produced by a Native Strain and in a Biochemical Reconstitution
- 下一个问题:这些化合物是天然产物还是表达产生的artifacts
- 利用两个实验证明,第一天然表达这两种BGC的菌株在不同的培养体系下面去测,看看能不能测到上面的小分子,这个方法验证了bgc52,但是没有验证bgc35
- 过表达bgc35 in Ecoli,然后纯化蛋白,体外重构了这个生物合成的通路,通过加底物,最后验证了2个compounds
The Gut NRPS Family Is Widely Distributed in Healthy Humans
- 下一个问题:这些cluster是否在人群样本中广泛分布?
- 用了两个方法去研究已有的宏基因组数据集做证明这些BGC的广泛分布:
- 针对这些BGC,用mBLASTx的方法去做map HMP
- 用了一个新的方法 gene catalog
- 用了两个方法去研究已有的宏基因组数据集做证明这些BGC的广泛分布:
Gut NRPS Clusters Are Actively Transcribed under Conditions of Host Colonization
- 针对有些BGC存在于metagenomic sample但是不表达或者表达很低的情况,为了去address gut NRPS是否在正常的人里面能够被转录,去分析了已有的人群的RNA-seq数据
The Active Gut NRPS Product May Be the Initially Released Dipeptide Aldehydes
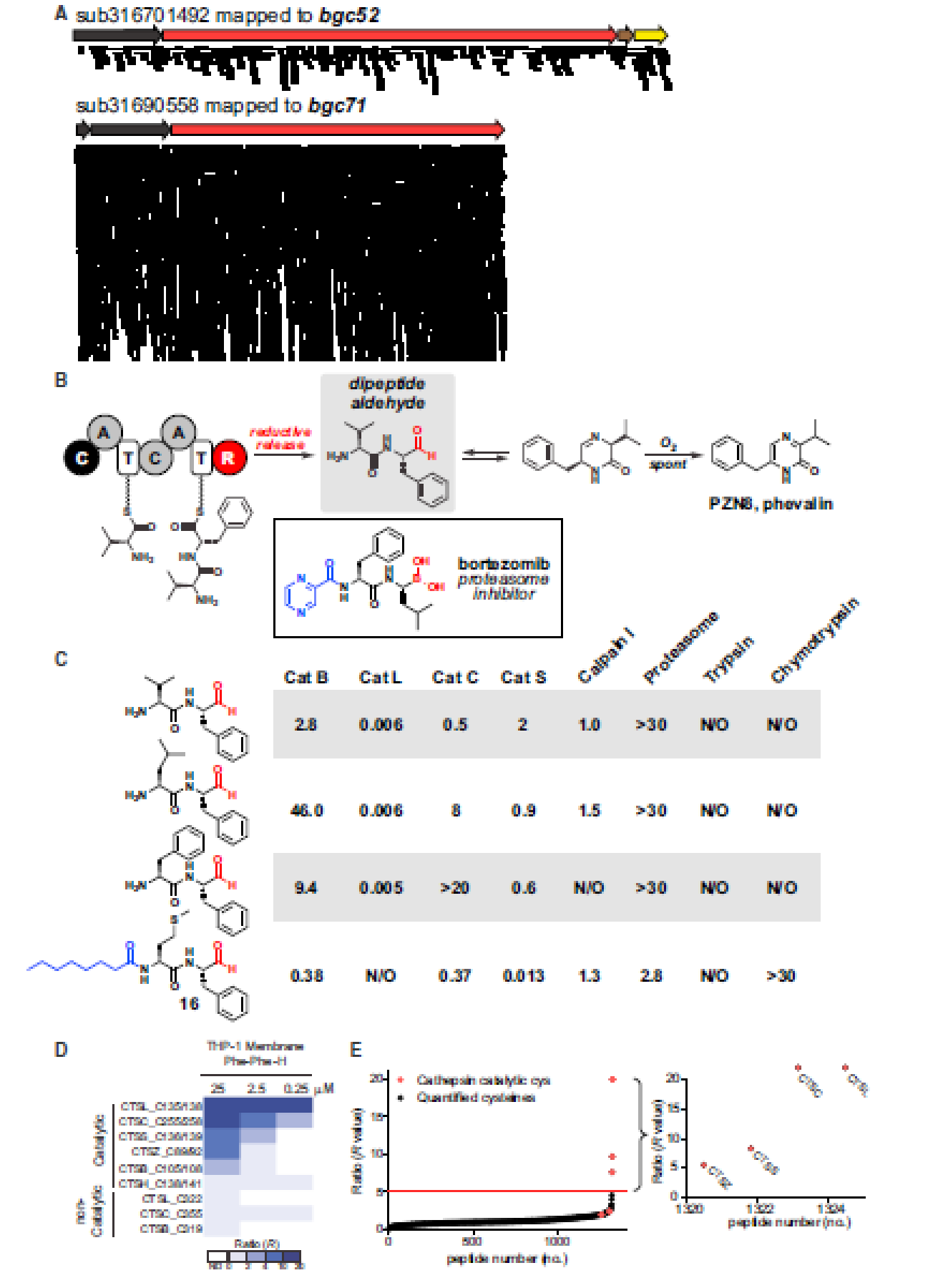
怎么猜出来这些是来源于dipeptide aldehydes没怎么看懂
-
猜测这些products是来源于dipeptide aldehydes,基于3个原因
- 测定了其中3个化合物的氧化半周期,在3-28h,表明有足够的时间在宿主中是稳定的,推导了前体Phe-Phe-H
- 有很久的文献历史表明peptide aldehydes是蛋白酶抑制剂
- in vitro实验测定了这几个化合物对于不同的蛋白酶的抑制活性
-
最后用isoTOP-ABPP方法Phe-Phe-H找到bgc35的靶点
感受
-
首先是文章思路。刚读这篇文章的时候觉得前面的思路可能就是follow 2014年那篇cell,但是后面发现和那篇cell相比,这篇文章多了明显更多的生化方面的研究,并且做了一些机制上的探究。从BGC出发,到鉴定MDM,再推测来源,发现dipeptide aldehydes,然后证明具备其具备蛋白酶抑制活性及其中一个的靶点
-
之前纯天然产物方面的文章接触的很少,读完这篇文章还是有很多启发。
- 对于未知物的鉴定。这篇文章先用大量体外的技术通过生化手段进行体外表达,然后通过分离纯化,结合LC紫外,LC-MSMS,以及核磁方面来共同鉴定MDM。这套方法应该是我要学习的方法,MetDNA2这个课题里面太拘泥于用纯bioinformatics的方法来发现,其实完全可以用文章中的这些方法来进行分离
- 鉴定到的unknown可以做的更加深入,比如这篇文章,从人群角度去探讨,从应用如抑制剂角度,靶点去做分析,做到更深的深度
-
需要学习的方法自然有很多,最相关的还是BGC那一套,以及思考有没有可能将非靶向代谢组的数据和BGC做联用。